預備
library(tidyverse)資料匯入與匯出
不同格式的檔案需要用相對應的 package 來處理匯入與匯出,比如 tidyverse 裡的 readr 只適用於 .txt 和 .csv 檔案。
readxl 可以處理 .xls 和 .xlsx 檔案。
數據可能是以不同的分隔符號紀錄在檔案裡,需要用相對應的函數來處理。比如最常見的三種是逗號(,)分隔,分號(;)分隔,以及 Tab 分隔。
# comma separated
import.data <- readr::read_csv("file_name.csv")
# semicolon separated
import.data <- readr::read_csv2("file_name.csv")
# tab separated
import.data <- readr::read_tsv("file_name.csv")以 CPS 資料為例,如果下載 .txt 格式。
# .txt file
cps <- read_tsv("cps09mar.txt", col_names = FALSE) # 匯入的資料並沒有變數名稱,且以 tab 分隔
write_csv(cps, "output.txt") # 匯出會自動新增變數名稱,並以逗號分隔
read_csv("output.txt") # 因為是逗號分隔,所以讀取資料時,改成用 read_csv()如果下載 .xlsx 格式。使用 readxl package 處理。
# install.packages("readxl")
library(readxl)
cps <- read_xlsx("cps09mar.xlsx")
write_csv(cps, "output.csv") # 匯出以 csv 格式儲存如果要儲存 R 跑完的資料,使用 save(),放物件名稱,並指定存入的檔名,小心副檔名不要打錯。
load() 指定讀入的檔案,可以觀察到 Environment 出現儲存的物件名稱。
df1 <- mpg
df2 <- msleep
df3 <- txhousing
save(df1, df2, df3,
file = "mydata.RData")
load("mydata.RData")缺失值
以 ggplot2::msleep 為例,有許多變數的觀測值並沒有被紀錄。
ggplot2::msleep#> # A tibble: 83 x 11
#> name genus vore order conservation sleep_total sleep_rem sleep_cycle awake
#> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 Cheet~ Acin~ carni Carn~ lc 12.1 NA NA 11.9
#> 2 Owl m~ Aotus omni Prim~ <NA> 17 1.8 NA 7
#> 3 Mount~ Aplo~ herbi Rode~ nt 14.4 2.4 NA 9.6
#> 4 Great~ Blar~ omni Sori~ lc 14.9 2.3 0.133 9.1
#> 5 Cow Bos herbi Arti~ domesticated 4 0.7 0.667 20
#> 6 Three~ Brad~ herbi Pilo~ <NA> 14.4 2.2 0.767 9.6
#> 7 North~ Call~ carni Carn~ vu 8.7 1.4 0.383 15.3
#> 8 Vespe~ Calo~ <NA> Rode~ <NA> 7 NA NA 17
#> 9 Dog Canis carni Carn~ domesticated 10.1 2.9 0.333 13.9
#> 10 Roe d~ Capr~ herbi Arti~ lc 3 NA NA 21
#> # ... with 73 more rows, and 2 more variables: brainwt <dbl>, bodywt <dbl>使用 dplyr::drop_na() 可以將有遺漏的數據整筆刪除。
df <- msleep %>% drop_na()
df#> # A tibble: 20 x 11
#> name genus vore order conservation sleep_total sleep_rem sleep_cycle awake
#> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 Great~ Blar~ omni Sori~ lc 14.9 2.3 0.133 9.1
#> 2 Cow Bos herbi Arti~ domesticated 4 0.7 0.667 20
#> 3 Dog Canis carni Carn~ domesticated 10.1 2.9 0.333 13.9
#> 4 Guine~ Cavis herbi Rode~ domesticated 9.4 0.8 0.217 14.6
#> 5 Chinc~ Chin~ herbi Rode~ domesticated 12.5 1.5 0.117 11.5
#> 6 Lesse~ Cryp~ omni Sori~ lc 9.1 1.4 0.15 14.9
#> 7 Long-~ Dasy~ carni Cing~ lc 17.4 3.1 0.383 6.6
#> 8 North~ Dide~ omni Dide~ lc 18 4.9 0.333 6
#> 9 Big b~ Epte~ inse~ Chir~ lc 19.7 3.9 0.117 4.3
#> 10 Horse Equus herbi Peri~ domesticated 2.9 0.6 1 21.1
#> 11 Europ~ Erin~ omni Erin~ lc 10.1 3.5 0.283 13.9
#> 12 Domes~ Felis carni Carn~ domesticated 12.5 3.2 0.417 11.5
#> 13 Golde~ Meso~ herbi Rode~ en 14.3 3.1 0.2 9.7
#> 14 House~ Mus herbi Rode~ nt 12.5 1.4 0.183 11.5
#> 15 Rabbit Oryc~ herbi Lago~ domesticated 8.4 0.9 0.417 15.6
#> 16 Labor~ Ratt~ herbi Rode~ lc 13 2.4 0.183 11
#> 17 Easte~ Scal~ inse~ Sori~ lc 8.4 2.1 0.167 15.6
#> 18 Thirt~ Sper~ herbi Rode~ lc 13.8 3.4 0.217 10.2
#> 19 Pig Sus omni Arti~ domesticated 9.1 2.4 0.5 14.9
#> 20 Brazi~ Tapi~ herbi Peri~ vu 4.4 1 0.9 19.6
#> # ... with 2 more variables: brainwt <dbl>, bodywt <dbl>也可以針對某些變數的缺失值做處理。
df2 <- msleep %>% drop_na(sleep_rem)
df2#> # A tibble: 61 x 11
#> name genus vore order conservation sleep_total sleep_rem sleep_cycle awake
#> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 Owl m~ Aotus omni Prim~ <NA> 17 1.8 NA 7
#> 2 Mount~ Aplo~ herbi Rode~ nt 14.4 2.4 NA 9.6
#> 3 Great~ Blar~ omni Sori~ lc 14.9 2.3 0.133 9.1
#> 4 Cow Bos herbi Arti~ domesticated 4 0.7 0.667 20
#> 5 Three~ Brad~ herbi Pilo~ <NA> 14.4 2.2 0.767 9.6
#> 6 North~ Call~ carni Carn~ vu 8.7 1.4 0.383 15.3
#> 7 Dog Canis carni Carn~ domesticated 10.1 2.9 0.333 13.9
#> 8 Goat Capri herbi Arti~ lc 5.3 0.6 NA 18.7
#> 9 Guine~ Cavis herbi Rode~ domesticated 9.4 0.8 0.217 14.6
#> 10 Grivet Cerc~ omni Prim~ lc 10 0.7 NA 14
#> # ... with 51 more rows, and 2 more variables: brainwt <dbl>, bodywt <dbl>符號意義
\(\sim\): is distributed as
\(\Phi(\cdot)\): 標準常態分配的累積機率函數
\(\Phi^{-1}(\cdot)\): 標準常態分配的 quantile 函數
note that:
\(\Phi(-c) = 1 - \Phi(c) = \alpha\)
\(c = -\Phi^{-1}(\alpha) = \Phi^{-1}(1 - \alpha)\)
\(T_n(\cdot)\): t 分配,自由度 n 的累積機率函數
\(T^{-1}_n(\cdot)\): t 分配,自由度 n 的 quantile 函數
note that:
\(T_n(-c) = 1 - T_n(c) = \alpha\)
\(c = -T_n^{-1}(\alpha) = T_n^{-1}(1 - \alpha)\)
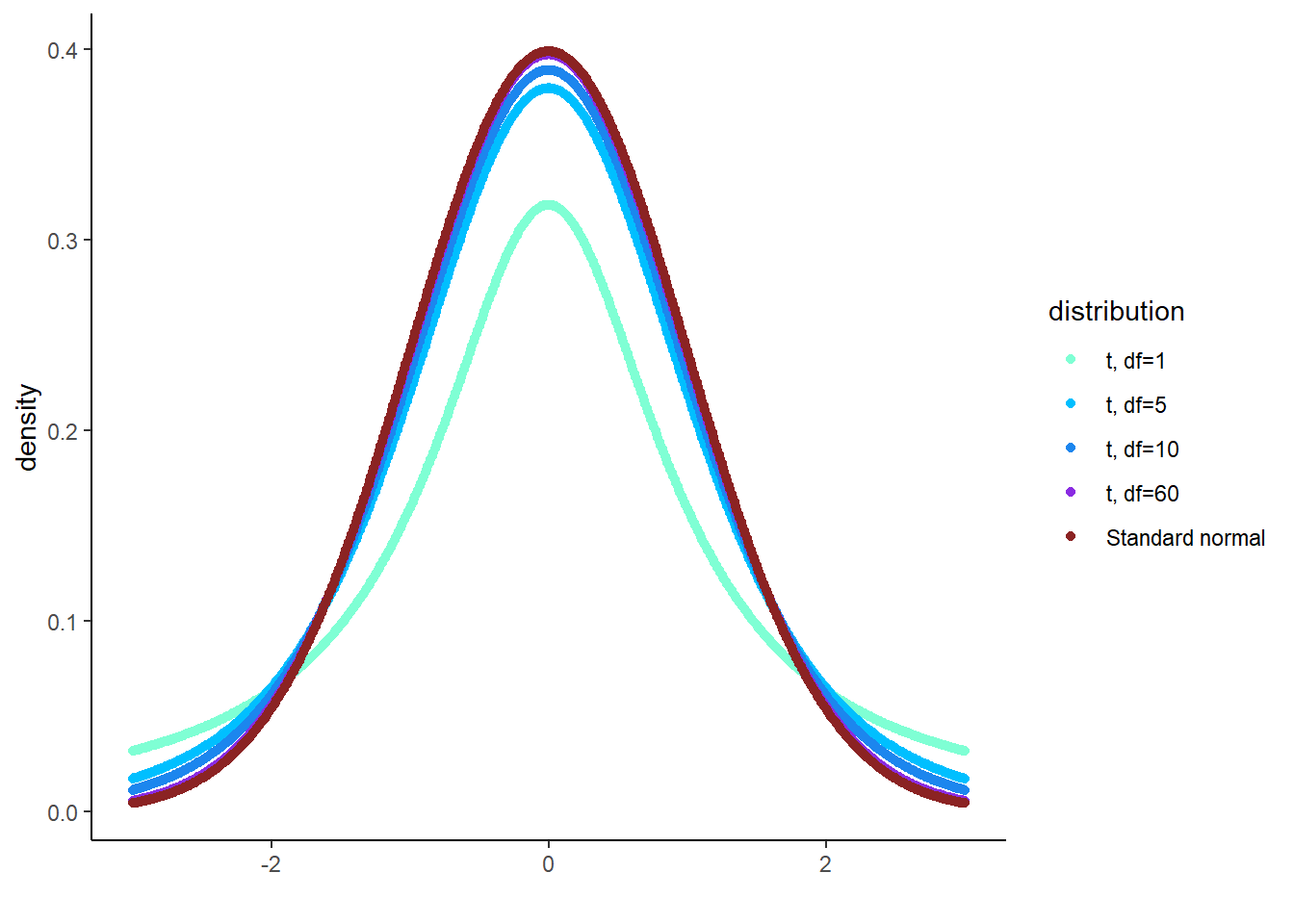
標準常態分配與 t 分配
信賴區間
- 單一母體平均數的區間估計
假設母體常態分配,以 \(95\%\) 信賴區間,雙尾檢定為例。
母體變異數已知
\[ \begin{aligned} &X_1, X_2,\dots, X_n \sim N(\mu, \sigma^2)\\\\ &\frac{\bar X_n - \mu}{\frac{\sigma}{\sqrt n}} \sim N(0,1)\\\\ &c = -\Phi^{-1}(0.025) = \Phi^{-1}(0.975)\\\\ &0.95 = P(-c < \frac{\bar X_n - \mu}{\frac{\sigma}{\sqrt n}} < c)\\\\ &0.95 = P(\bar X_n - c\frac{\sigma}{\sqrt{n}} < \mu < \bar X_n + c\frac{\sigma}{\sqrt{n}}) \end{aligned} \]
在母體常態分配,平均值為 0, 標準差為 2,隨機抽出 100 個樣本,找出此次抽樣的信賴區間,並畫圖表示。
# 隨機抽樣
random.sample = rnorm(n = 100, mean = 0, sd = 2)
# 樣本平均數
sample.mean <- mean(random.sample)
sample.mean#> [1] 0.005825125# 標準常態分配 2.5% 和 97.5% 的位置
z_0.025 <- qnorm(0.025, mean = 0, sd = 1)
z_0.975 <- qnorm(0.975, mean = 0, sd = 1)
round(c(z_0.025, z_0.975), 2)#> [1] -1.96 1.96const <- z_0.975
# 信賴區間
lower <- sample.mean - const * 2 / sqrt(100)
upper <- sample.mean + const * 2 / sqrt(100)
round(c(lower, upper), 4)#> [1] -0.3862 0.3978# 畫圖
## 法一
ggplot() +
geom_point(aes(random.sample, dnorm(random.sample, 0, 2))) +
geom_vline(xintercept = c(lower, upper),
linetype = "dashed",
color = "red")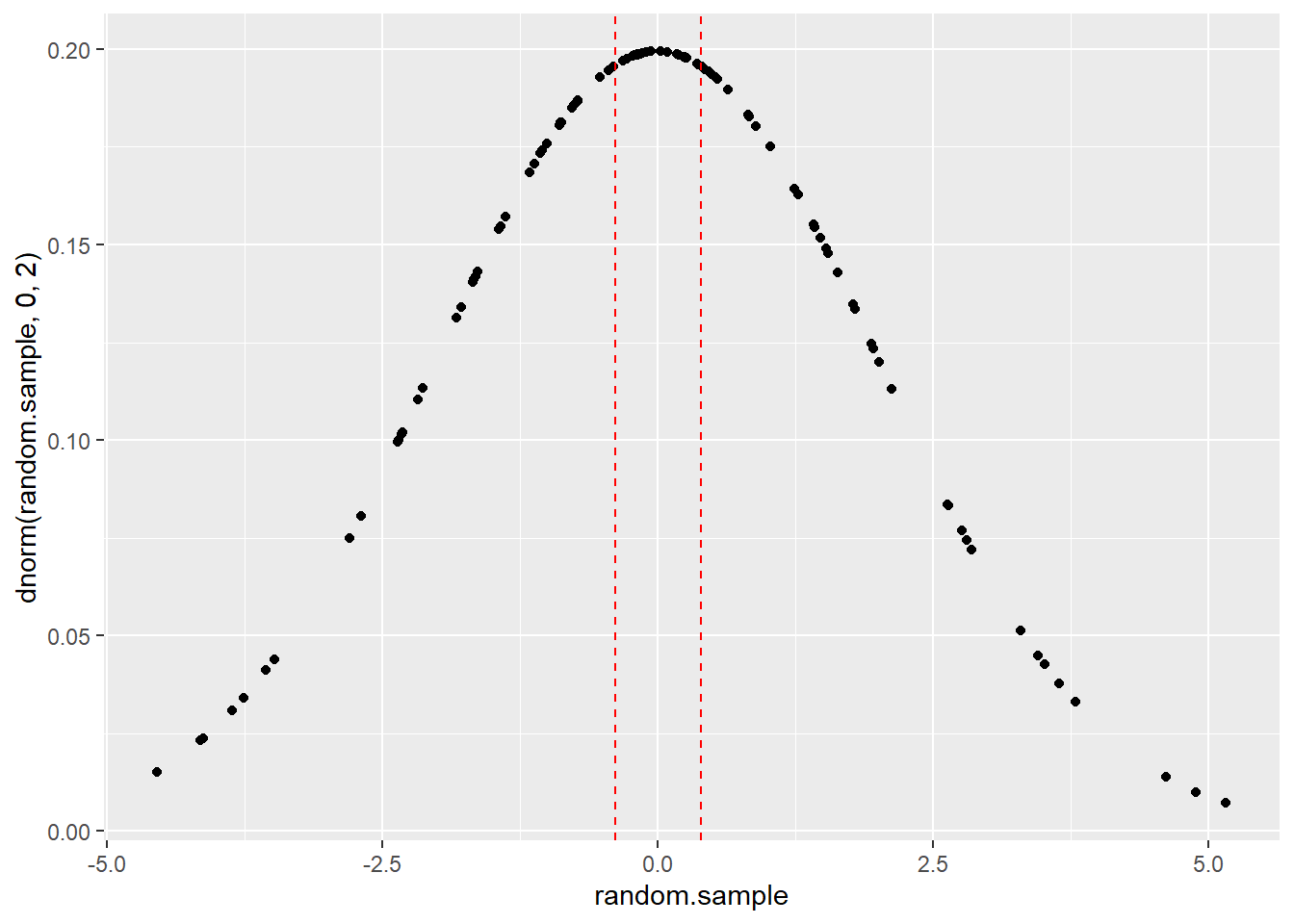
## 法二
data <- tibble(x = random.sample,
y = dnorm(random.sample, 0, 2))
ggplot(data) +
geom_point(aes(x, y)) +
geom_vline(xintercept = c(lower, upper),
linetype = "dashed",
color = "red",
size = 1)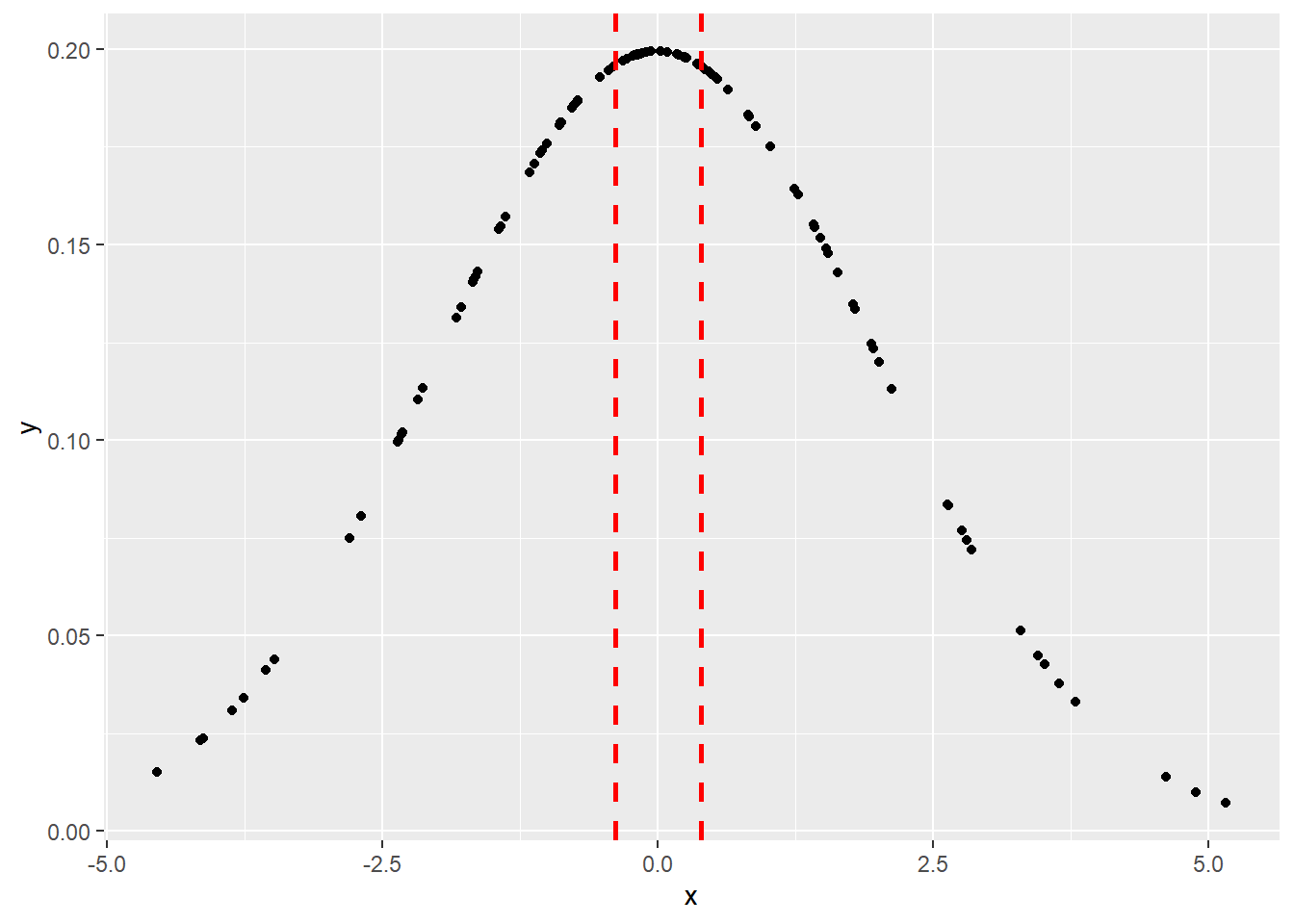
## 法三
data <- tibble(x = rnorm(100, 0, 2),
y = dnorm(x, 0, 2))
ggplot(data) +
geom_point(aes(x, y)) +
geom_vline(xintercept = c(mean(data$x) - const * 2 / sqrt(100),
mean(data$x) + const * 2 / sqrt(100)),
linetype = "dashed",
color = "red",
size = 1)母體變異數未知
\[ \begin{aligned} &X_1, X_2,\dots, X_n \sim N(\mu, \sigma^2)\quad S_n^2 = \frac{1}{n-1}\sum(X_i-\bar X_n)^2\\\\ &\frac{\bar X_n - \mu}{\frac{S_n}{\sqrt n}} \sim t_{n-1}\\\\ &c = -T_{n-1}^{-1}(0.025) = T_{n-1}^{-1}(0.975)\\\\ &0.95 = P(-c < \frac{\bar X_n - \mu}{\frac{S_n}{\sqrt n}} < c)\\\\ &0.95 = P(\bar X_n - c\frac{S_n}{\sqrt{n}} < \mu < \bar X_n + c\frac{S_n}{\sqrt{n}}) \end{aligned} \]
在母體常態分配,平均值為 0, 標準差未知,隨機抽出 100 個樣本,找出此次抽樣的信賴區間,並畫圖表示。
# 隨機抽樣
random.sample = rnorm(n = 100, mean = 0, sd = 2)
# 樣本平均數
sample.mean <- mean(random.sample)
sample.mean#> [1] 0.02228167# S_n
S_n <- sqrt(sum((random.sample - sample.mean) ** 2) / (100 - 1))
S_n#> [1] 1.592378# t 分配 2.5% 和 97.5% 的位置
t_0.025 <- qt(0.025, df = 100 - 1)
t_0.975 <- qt(0.975, df = 100 - 1)
round(c(t_0.025, t_0.975), 2)#> [1] -1.98 1.98const <- t_0.975
# 信賴區間
lower <- sample.mean - const * S_n / sqrt(100)
upper <- sample.mean + const * S_n / sqrt(100)
round(c(lower, upper), 2)#> [1] -0.29 0.34# 畫圖
## 法一
ggplot() +
geom_point(aes(random.sample, dnorm(random.sample, 0, 2))) +
geom_vline(xintercept = c(lower, upper),
linetype = "dashed",
color = "red")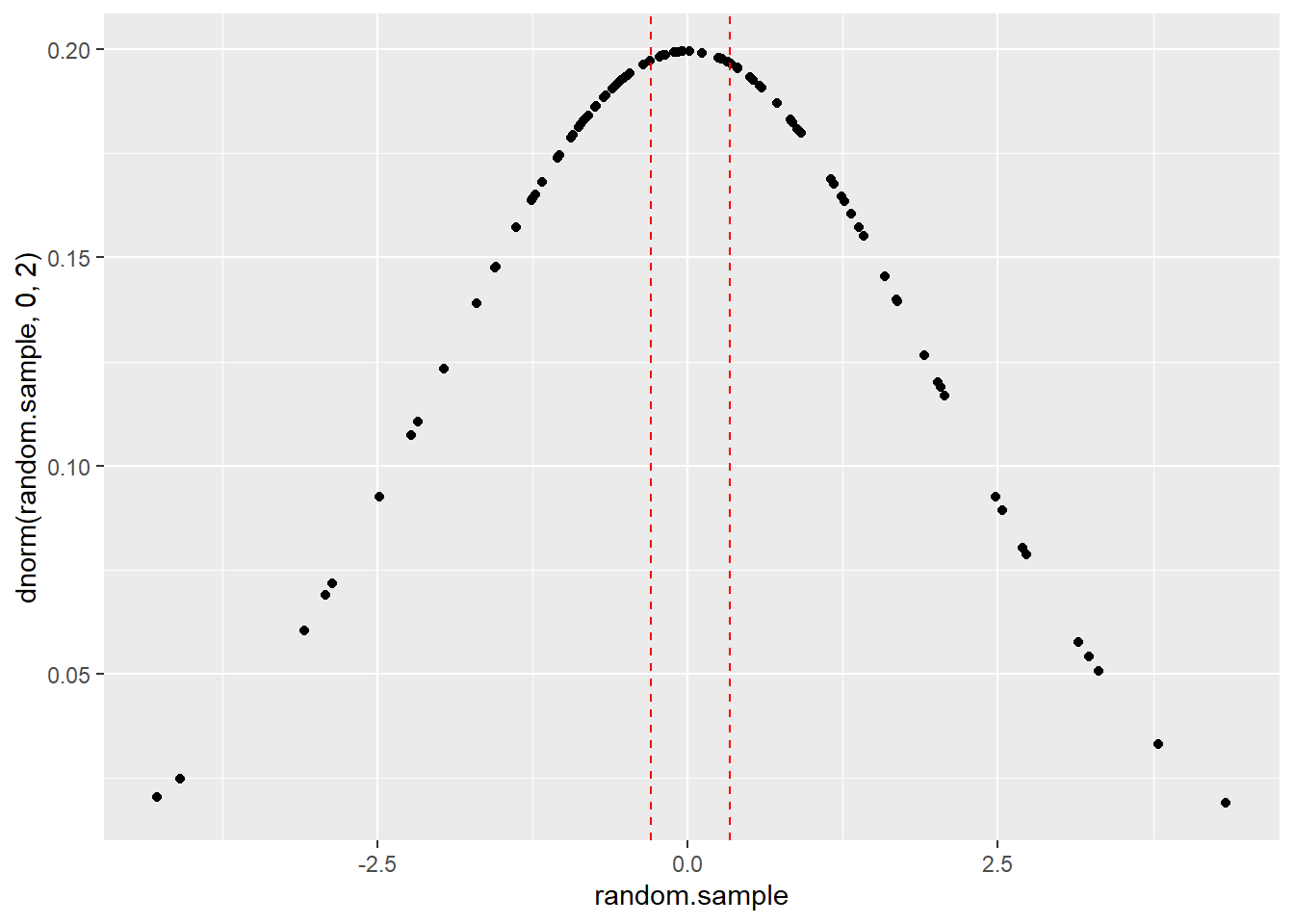
驗證
模擬重複抽樣 100 次,每次抽樣均抽出 100 組樣本,驗證雙尾檢定下 95% 的信賴區間。
在觀察到樣本前,預期樣本的信賴區間有 95% 的可能涵蓋母體平均數, 我們檢驗在重複抽樣 100 次後,有多少次抽樣的信賴區間包含母體平均數。
# 模擬
simulate <- map(1:100, ~ tibble(r = rnorm(100, 0, 2)))
lower_upper <- simulate %>%
map(~ tibble(lower = mean(.x$r) - qnorm(0.975, 0, 1) * 2 / sqrt(100),
upper = mean(.x$r) - qnorm(0.025, 0, 1) * 2 / sqrt(100))) %>%
bind_rows()
lower_upper#> # A tibble: 100 x 2
#> lower upper
#> <dbl> <dbl>
#> 1 -0.366 0.418
#> 2 -0.558 0.226
#> 3 -0.655 0.129
#> 4 -0.144 0.640
#> 5 -0.518 0.266
#> 6 -0.0542 0.730
#> 7 0.0739 0.858
#> 8 -0.605 0.179
#> 9 -0.273 0.511
#> 10 -0.553 0.231
#> # ... with 90 more rowslower_upper %>%
mutate(row = row_number()) %>%
ggplot() +
geom_segment(aes(x = lower, y = row,
xend = upper, yend = row),
color = "violetred4",
size = 1) +
labs(x = "confidence interval",
y = "simulation")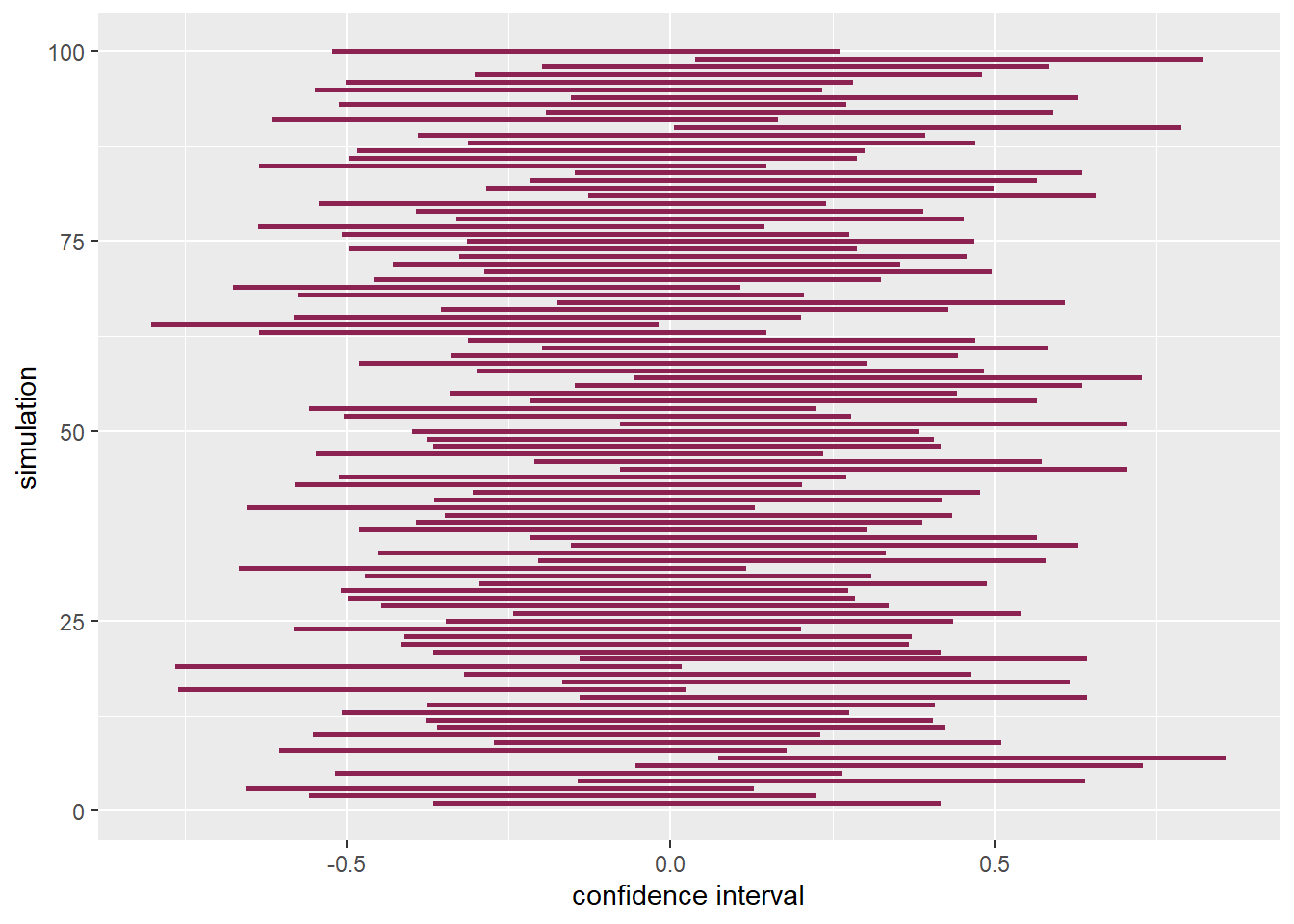
confidence <- lower_upper %>%
mutate(cover =
case_when(lower > 0 | upper < 0 ~ 0,
TRUE ~ 1)) %>%
summarise(cover = sum(cover))
confidence#> # A tibble: 1 x 1
#> cover
#> <dbl>
#> 1 96本次模擬的結果有 96% 的信賴區間涵蓋母體平均數。
作業
請應用區間估計對資料的變數做說明。
補充
- 單一母體變異數的區間估計
假設母體常態分配,以 \(95\%\) 信賴區間,雙尾檢定為例。
Hint: \(\chi^2_n\) 系列的函數為 *chisq()。
母體平均數已知
\[ \begin{aligned} &X_1, X_2,\dots, X_n \sim N(\mu, \sigma^2)\\\\ &\frac{\sum(X_i - \mu)^2}{\sigma^2} \sim \chi^2_{n}\\\\ &0.95 = P(\chi^2_{n, 0.025} < \frac{\sum(X_i - \mu)^2}{\sigma^2} < \chi^2_{n, 0.975})\\\\ &0.95 = P(\frac{\sum(X_i - \mu)^2}{\chi^2_{n, 0.975}} < \sigma^2 < \frac{\sum(X_i - \mu)^2}{\chi^2_{n, 0.025}}) \end{aligned} \]
母體平均數未知
\[ \begin{aligned} &X_1, X_2,\dots, X_n \sim N(\mu, \sigma^2)\\\\ &\frac{\sum(X_i - \bar X_n)^2}{\sigma^2} \sim \chi^2_{n-1}\\\\ &0.95 = P(\chi^2_{n-1, 0.025} < \frac{\sum(X_i - \bar X_n)^2}{\sigma^2} < \chi^2_{n-1, 0.975})\\\\ &0.95 = P(\frac{\sum(X_i - \bar X_n)^2}{\chi^2_{n-1, 0.975}} < \sigma^2 < \frac{\sum(X_i - \bar X_n)^2}{\chi^2_{n-1, 0.025}}) \end{aligned} \]